 Sven Saupe, Institut de Biochimie et Génétique Cellulaire, UMR 5095
Sven Saupe, Institut de Biochimie et Génétique Cellulaire, UMR 5095
Je suis directeur de recherche CNRS, généticien à l'IBGC (Institut de Biochimie et Génétique Cellulaire UMR 5095) et responsable de l'équipe " Non-Self Recognition in Fungi ". Nous nous intéressons aux mécanismes régissant la reconnaissance du non-soi dans cette vaste branche de l'arbre eucaryote. Nous étudions divers aspects de la détection et de la réponse au non-soi chez les champignons, avec un accent particulier sur une famille de récepteurs cytoplasmiques appelés NLR (Nod-like receptors) qui contrôlent l'immunité innée et d'autres processus d'interaction biotique dans les lignées animales et végétales. Les protéines NLR sont des récepteurs intracellulaires qui constituent un composant conservé du système immunitaire inné des organismes cellulaires. Chez les champignons, les NLR sont caractérisés par une grande diversité d'architectures et la présence d'une signalisation amyloïde.
Les amyloïdes sont impliqués dans de nombreuses maladies dues à un mauvais repliement des protéines (maladies d'Alzheimer et de Parkinson et diabète de type 2). Les amyloïdes fonctionnels impliqués dans le contrôle des mécanismes de régulation de la mort cellulaire n'ont été caractérisés jusqu'à présent que chez les champignons et les mammifères. Le projet Infranalytics visait à caractériser une amyloïde fonctionnelle impliquée dans l'activation du NLR chez les bactéries. Une méthode de choix pour étudier les amyloïdes au niveau atomique est la résonance magnétique nucléaire (RMN) à l'état solide. Le développement de stratégies de marquage isotopique a considérablement amélioré la puissance de la RMN des solides dans les études structurales, car les schémas de marquage permettent de récupérer sélectivement les informations souhaitées (c'est-à-dire une position spécifique des noyaux marqués 13C et 15N dans les protéines amyloïdes). Ce projet a été développé en collaboration avec Antoine Loquet.
Nous avons identifié un certain nombre de motifs de signalisation amyloïde bactérienne dans les génomes bactériens que nous appelons « tem BASS » (bacterial amyloid signaling sequence). Au cours de ce projet Infranalytics, nous avons utilisé la RMN à l'état solide pour attribuer le noyau rigide de la protéine BASS1 dans son état fibrillaire.
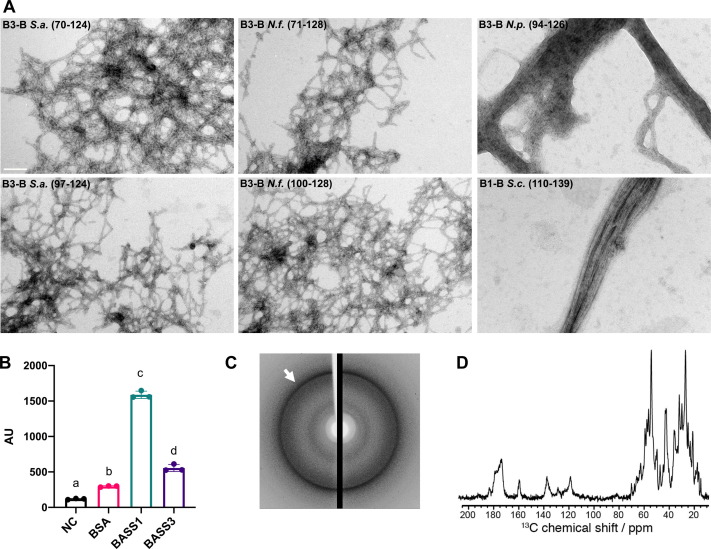
En savoir plus : https://doi.org/10.1016/j.jmb.2020.10.004
Plateforme utilisée : CBMN - IECB (Bordeaux)
