 Patrick AUGUSTE, chercheur au sein de l'UMR 8198 Evolution, Ecologie et Paléontologie (EEP), CNRS - Université de Lille
Patrick AUGUSTE, chercheur au sein de l'UMR 8198 Evolution, Ecologie et Paléontologie (EEP), CNRS - Université de Lille
Je suis paléontologue et archéozoologue, je travaille sur les fossiles de mammifères découverts dans les sites paléolithiques. Mes objectifs sont d’identifier les espèces animales présentes dans les accumulations laissés par les Hommes préhistoriques, afin d’étudier l’évolution de la biodiversité passée, les changements environnementaux, mais également les comportements de subsistance des différentes populations d’Hominidés responsables des abattages de ces animaux. Je suis ainsi amené à devoir identifier de très petits fragments d’ossements, liés à la fracturation des os afin de récupérer la moelle.
Une collaboration s’est établie avec l’équipe des chimistes de MSAP, afin de développer de nouveaux protocoles d’extraction de matière osseuse d’ossements anciens à très anciens (40 000 à 600 000 ans) et d’identifier les différentes espèces animales rencontrées lors des fouilles archéologiques et paléontologiques. Nous nous sommes tournés vers le Maldi-FT-ICR, de par sa très haute résolution et la moindre quantité de matière à prélever pour chaque échantillon.
Patrick Auguste qui tient un crâne de lion des cavernes, avec en arrière plan des pyrogravures d'un ours de cavernes et d'un félin à dent de sabre
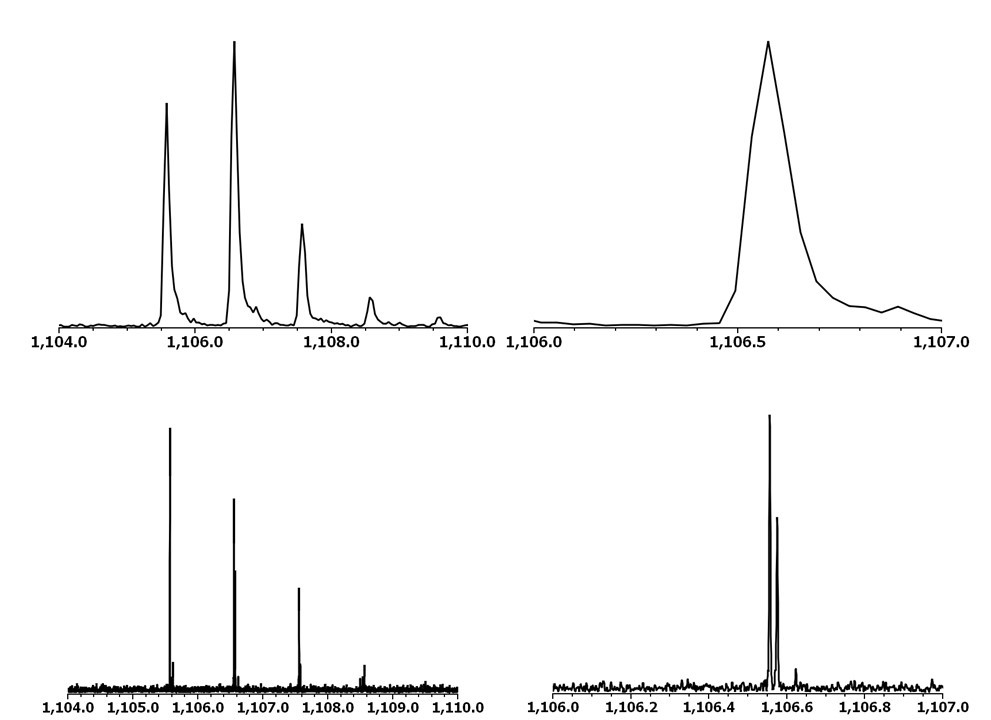
Comparaison entre des spectres obtenus par Maldi-Tof et Maldi-FTICR sur du collagène d'aurochs de 123 000 ans
Cette collaboration interdisciplinaire, très fortement soutenue par le CNRS, la région Hauts-de-France et l’ANR, nous a ainsi permis de perfectionner considérablement la discipline paléoprotéomique selon deux axes :
1/ diminuer la quantité d’os prélevé dans chaque échantillon, en passant à 1 mg (les autres méthodes utilisent de 5 à 100 mg)
2/ augmenter le nombre d’échantillons traités par analyse FT-ICR (plaques de 96 puits).
Un test en grandeur nature a été réalisé sur des os datés de 123 000 ans, très peu identifiables par l’ostéomorphologie, et 93 % des 104 os échantillonnés ont permis une identification des animaux, objet de l’article récemment paru.
En savoir plus : https://doi.org/10.1021/acs.analchem.2c03301.
Plateforme utilisée : MSAP, CNRS/Université de Lille
