Le Centre de RMN à Très Hauts Champs (CRMN) est une unité mixte de recherche du CNRS, de l'Université Claude Bernard de Lyon 1 et de l'Ecole Normale Supérieure de Lyon. Les équipes du CRMN travaillent sur les aspects les plus fondamentaux de la RMN, à l'interface de la biologie, de la chimie et de la physique, afin de i) développer des percées méthodologiques et instrumentales, ii) étendre radicalement le champ d'application de cette spectroscopie, et iii) permettre l'exploration de la structure et de la dynamique de nouvelles gammes de composés à l'échelle atomique dans des domaines scientifiques clés ayant un impact sociétal significatif.
Le centre a été conçu autour de l'installation du premier spectromètre mondial à 1 GHz, livré à l'été 2009 et progressivement équipé d'une variété d'accessoires de pointe (y compris des sondes pour la rotation à l'angle magique ultra-rapides et des cryosondes pour la solution). Autour de cet instrument, une plateforme a été constituée de six autres spectromètres de fréquence 400, 500, 600, 700 et 800 (x2) MHz, offrant des capacités de recherche exceptionnelles. Aujourd'hui, le centre héberge en outre une plateforme unique de RMN hyperpolarisée avec deux systèmes MAS DNP à 400 MHz et 800 MHz, ainsi que deux polariseurs pour la DNP par dissolution.
- Expertises et services
- RMN biologique en solution (structure, dynamique, intéractions des protéines ou acides nucléiques)
- RMN biologique à l'état solide avec rotation ultra-rapide de l'échantillon à l'angle magique (protéines membranaires, fibrilles amyloïdes, assemblages biologiques, ...)
- MAS DNP pour l'étude des surfaces (catalyseurs hétérogènes, composés adsorbés, ...)
- RMN des matériaux à l'état solide avec rotation de l'échantillon à l'angle magique (catalyseurs, polymères, ...)
- RMN paramagnétique
- Autres équipements
800 MHz Bruker AVANCE NEO
• 5 sondes RMN solide: 0.7mm HCN, 1.3mm HCN, 1.3mm HCND, 2.5mm BBHX, 3.2mm HCN
• 1 sonde RMN liquide : TXI 5mm
• SampleJet autosampler (5 racks de 96 échantillons et 30 spinners)
• 1 sonde RMN HR-MAS
700 MHz Bruker AVANCE III
• 4 sondes RMN solide: 1.3mm HCN, 2.5mm BBHX, 3.2mm HCN, 4mm BBHX
• 2 sondes RMN liquide : TXI 5mm, BBO 5mm
• 1 sonde RMN HR-MAS
• HR-MAS SamplePro autosampler (50 échantillons)
600 MHz Bruker AVANCE III
• 2 cryosondes 5mm : HCND, H/FPCN
• SampleJet autosampler (5 racks de 96 échantillons et 30 spinners)
500 MHz Bruker AVANCE NEO
• 9 sondes RMN solide : 0.7mm HXY, 1.3mm HX, 2.5mm BBHX, 2.5mm CRAMPS, 3.2mm HCN, 4mm HXY, 4mm BB HX, 7mm BB HX, Static HX
• Cryocabinet LT-MAS
400 MHz Bruker AVANCE III HD
• Gyrotron 263 GHz
• Cryocabinet LT-MAS
• 2 sondes RMN DNP : 1.3mm LTMAS HXY, 3.2mm LTMAS HXY
Prototype polariseur d-DNP 300 MHz / 7.05 Tesla / 198 GHz Bruker AVANCE III HD
• Cabinet de DNP par dissolution (d-DNP)
• Cryostat 1.2K
• 2 sondes RMN 1H/13C, 1H/15N


La plateforme Infranalytics de Gif-sur-Yvette comprend :

 • Un spectromètre 950 MHz Bruker Avance NEO équipé d'une cryosonde TCI (1H,13C,15N,2H) 5mm, gradients-z (S/B = 9307) travaillant de 0°C à 80°C, compatible tubes ovales (échantillons salins)
• Un spectromètre 950 MHz Bruker Avance NEO équipé d'une cryosonde TCI (1H,13C,15N,2H) 5mm, gradients-z (S/B = 9307) travaillant de 0°C à 80°C, compatible tubes ovales (échantillons salins)• Un équipement « Deadalus inc» pour réaliser des expériences de RMN à haute pression, avec 1 tube en céramique (1 bar à 3 kbar)
Expériences RMN disponibles :
RMN rapide (BEST, SOFAST, ...), Méthodes d’échantillonnage non linéaire ou de projection, Expérience triple résonance 3D, 4D pour protéines et acides nucléiques, Relaxation et Dispersion de Relaxation, Couplages Dipolaires Résiduels, RMN des méthyls, Interactions Protéines ligands, DOSY, RMN paramagnétique (PRE/PCS).Domaines principaux d'application :
Structure, dynamique et interactions des macromolécules biologiques ; petites molécules en faible quantité (produits naturels).Resp. Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser. (33-(0)169823764) ; Corr. Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser. (33-(0)169823762) ; Secr-gestion Cette adresse e-mail est protégée contre les robots spammeurs. Vous devez activer le JavaScript pour la visualiser. (33-(0)169823671)
- Expertises et services
- Conception de projets d’étude structurale par RMN adaptés aux systèmes et questions posées et accompagnement des équipes à chaque étape : attribution de résonances, étude de dynamique, détermination de structures 3D, interactions, titrages, cinétiques...
- Réalisation d’expériences de RMN multi-noyaux (1H, 2H, 13C, 15N, 19F, 31P), RMN rapide (BEST, SOFAST, ...), RMN paramagnétique et haute pression
- Utilisation de méthodes d’échantillonnage non linéaire ou de projections
- Réalisation d’expériences triple résonance 3D et 4D pour protéines et acides nucléiques
- Réalisation d’expériences de relaxation, dispersion de relaxation, couplage dipolaire résiduel et diffusion (DOSY)
- Enregistrement de spectres RMN spécifiques des méthyls
- Enregistrement d’expériences RMN pour la caractérisation d’interactions protéine-ligand
- Analyse et interprétation de données avec formation des utilisateurs
- Mise à disposition de spectromètres avec accompagnement pour utilisateurs avertis.
- Autres équipements

Le spectromètre 950MHz est intégré dans la plateforme RMNHC@UPSAY, noeud RMN de l'infrastructure FRISBI (French Research Integrated Structural Biology Infrastructure), labélisée IBISA, dont 5 des 6 spectromètres sont actuellement gérés par l’équipe "RMN, Biologie et Chimie Structurales" de l’Institut de Chimie des Substances Naturelles, sur le campus CNRS de Gif-sur-Yvette. La recherche sur le campus est essentiellement centrée sur la chimie et la biologie, avec une forte composante de biologie structurale dans un contexte biologie et santé. Le campus de Gif est intégré dans le « grand campus Paris-Saclay » (https://www.universite-paris-saclay.fr/), qui regroupe plus de 10000 chercheurs de tous les horizons scientifiques, et une forte communauté de chercheurs en biologie structurale (Gif-sur-Yvette, Université Paris 11, le CEA à Saclay et le synchrotron SOLEIL).
800 MHz Bruker Avance III Liquide
- Sonde liquide:
- Cryo TCI (1H, 13C, 15N, 2H), 5mm, z-gradients
700 MHz Bruker Avance NEO Liquide
Sonde liquide TXO (1H, 13C, 15N, 2H), 5mm, z-gradients
600 MHz (Bruker-1) Bruker Avance III Liquide
Sondes liquides:
- Cryo TCI (1H, 13C, 15N, 2H), 5mm, z-gradients
- Cryo QCI (1H, 13C, 15N, 19F, 2H), 5mm, z-gradients
- Passeur d’échantillon SampleJet (avec régulation de température)
600 MHz (Bruker-2) Bruker Avance III Liquide
Sondes liquides:
- TXI (1H, 13C, 15N, 2H), 5mm, x,y,z-gradients
- TBI (1H, 13C BB), 5mm, x,y,z-gradients
De plus, un service de RMN est à la disposition des chimistes, avec un 600MHz, deux 500MHz et deux 300MHz.
Quelques publications récentes
Exploration of the dynamic interplay between lipids and membrane proteins by hydrostatic pressure.
Pozza A, Giraud F, Cece Q, Casiraghi M, Point E, Damian M, Le Bon C, Moncoq K, Banères J-L, Lescop E, Catoire L J. Nat Commun. 2022 Apr 1;13(1):1780. doi: 10.1038/s41467-022-29410-5. Online ahead of print. PMID: 35365643
Optimisation of spatially-encoded diffusion-ordered NMR spectroscopy for the analysis of mixtures.
Jacquemmoz C, Mishra R, Guduff L, van Heijenoort C, Dumez JN. Magn Reson Chem. 2022 Jan;60(1):121-138. doi: 10.1002/mrc.5194. Epub 2021 Sep 2. PMID: 34269476
Doyen C, Larquet E, Coureux PD, Frances O, Herman F, Sablé S, Burnouf JP, Sizun C, Lescop E. Mol Pharm. 2021 Jul 5;18(7):2521-2539. doi: 10.1021/acs.molpharmaceut.1c00037. Epub 2021 Jun 20. PMID: 34151567
Malard F, Jacquet E, Nhiri N, Sizun C, Chabrier A, Messaoudi S, Dejeu J, Betzi S, Zhang X, Thureau A, Lescop E. ChemMedChem. 2022 Jan 5;17(1):e202100528. doi: 10.1002/cmdc.202100528. Epub 2021 Oct 6. PMID: 344727
Investigation of the Low-Populated Excited States of the HIV-1 Nucleocapsid Domain.
Mouhand A, Zargarian L, Belfetmi A, Catala M, Pasi M, Lescop E, Tisné C, Mauffret O. Viruses. 2022 Mar 18;14(3):632. doi: 10.3390/v14030632. PMID: 35337039
Decool H, Gonnin L, Gutsche I, Sizun C, Eléouët JF, Galloux M. Viruses. 2021 Dec 6;13(12):2449. doi: 10.3390/v13122449. PMID: 34960719
Expanding the Disorder-Function Paradigm in the C-Terminal Tails of Erbbs.
Pinet L, Assrir N, van Heijenoort C. Biomolecules. 2021 Nov 14;11(11):1690. doi: 10.3390/biom11111690. PMID: 34827688
Relationships between RNA topology and nucleocapsid structure in a model icosahedral virus.
Marichal L, Gargowitsch L, Rubim RL, Sizun C, Kra K, Bressanelli S, Dong Y, Panahandeh S, Zandi R, Tresset G. Biophys J. 2021 Sep 21;120(18):3925-3936. doi: 10.1016/j.bpj.2021.08.021. Epub 2021 Aug 19. PMID: 34418368
β-Hairpin Peptide Mimics Decrease Human Islet Amyloid Polypeptide (hIAPP) Aggregation.
Lesma J, Bizet F, Berardet C, Tonali N, Pellegrino S, Taverna M, Khemtemourian L, Soulier JL, van Heijenoort C, Halgand F, Ha-Duong T, Kaffy J, Ongeri S. Front Cell Dev Biol. 2021 Sep 16;9:729001. doi: 10.3389/fcell.2021.729001. eCollection 2021. PMID: 34604227
Cardone C, Caseau CM, Bardiaux B, Thureaux A, Galloux M, Bajorek M, Eléouët JF, Litaudon M, Bontems F, Sizun C. Biomolecules. 2021 Aug 17;11(8):1225. doi: 10.3390/biom11081225. PMID: 34439894
Guérineau M, Bessa L, Moriau S, Lescop E, Bontems F, Mathy N, Guittet E, Bischerour J, Bétermier M, Morellet N. Mob DNA. 2021 Apr 29;12(1):12. doi: 10.1186/s13100-021-00240-4. PMID: 33926516
Structural and dynamic characterization of the C-terminal tail of ErbB2: Disordered but not random.
Pinet L, Wang YH, Deville C, Lescop E, Guerlesquin F, Badache A, Bontems F, Morellet N, Durand D, Assrir N, van Heijenoort C. Biophys J. 2021 Mar 17:S0006-3495(21)00234-4. doi: 10.1016/j.bpj.2021.03.005. Online ahead of print. PMID: 33741354
Drimane Derivatives as the First Examples of Covalent BH3 Mimetics that Target MCL-1.
Daressy F, Malard F, Seguy L, Guérineau V, Apel C, Dumontet V, Robert A, Groo AC, Litaudon M, Bignon J, Desrat S, Malzert-Fréon A, Wiels J, Lescop E, Roussi F. ChemMedChem. 2021 Mar 5. doi: 10.1002/cmdc.202100011. Online ahead of print. PMID: 33665938
Cardone C, Caseau CM, Pereira N, Sizun C. Int J Mol Sci. 2021 Feb 3;22(4):1537. doi: 10.3390/ijms22041537. PMID: 33546457 Free PMC article. Review.
Fluorinated Triazole Foldamers: Folded or Extended Conformational Preferences.
Laxio Arenas J, Xu Y, Milcent T, Van Heijenoort C, Giraud F, Ha-Duong T, Crousse B, Ongeri S. Chempluschem. 2021 Feb;86(2):241-251. doi: 10.1002/cplu.202000791. PMID: 33555641
Bajorek M, Galloux M, Richard CA, Szekely O, Rosenzweig R, Sizun C, Eleouet JF. J Virol. 2021 Jan 6:JVI.02217-20. doi: 10.1128/JVI.02217-20. Online ahead of print. PMID: 33408180
Chevreuil M, Lecoq L, Wang S, Gargowitsch L, Nhiri N, Jacquet E, Zinn T, Fieulaine S, Bressanelli S, Tresset G. J Phys Chem B. 2020 Nov 12;124(45):9987-9995. doi: 10.1021/acs.jpcb.0c05024. Epub 2020 Nov 2. PMID: 33135897
Munoz-Montesino C, Larkem D, Barbereau C, Igel-Egalon A, Truchet S, Jacquet E, Nhiri N, Moudjou M, Sizun C, Rezaei H, Béringue V, Dron M. J Biol Chem. 2020 Oct 9;295(41):14025-14039. doi: 10.1074/jbc.RA120.014738. Epub 2020 Aug 11. PMID: 32788216 Free article.
Cyclodextrin complexation studies as the first step for repurposing of chlorpromazine.
Wang Z, Landy D, Sizun C, Cézard C, Solgadi A, Przybylski C, de Chaisemartin L, Herfindal L, Barratt G, Legrand FX. Int J Pharm. 2020 Jun 30;584:119391. doi: 10.1016/j.ijpharm.2020.119391. Epub 2020 May 4. PMID: 32376444
Rivière G, Oueslati S, Gayral M, Créchet JB, Nhiri N, Jacquet E, Cintrat JC, Giraud F, van Heijenoort C, Lescop E, Pethe S, Iorga BI, Naas T, Guittet E, Morellet N. ACS Omega. 2020 Apr 28;5(18):10466-10480. doi: 10.1021/acsomega.0c00590. eCollection 2020 May 12. PMID: 32426604 Free PMC article.
Ubiquitin is double-phosphorylated by PINK1 for enhanced pH-sensitivity of conformational switch.
Ye SX, Gong Z, Yang J, An YX, Liu Z, Zhao Q, Lescop E, Dong X, Tang C. Protein Cell. 2019 Dec;10(12):908-913. doi: 10.1007/s13238-019-0644-x. PMID: 31278634 Free PMC article. No abstract available.
Defects in t6A tRNA modification due to GON7 and YRDC mutations lead to Galloway-Mowat syndrome.
Arrondel C, Missoury S, Snoek R, Patat J, Menara G, Collinet B, Liger D, Durand D, Gribouval O, Boyer O, Buscara L, Martin G, Machuca E, Nevo F, Lescop E, Braun DA, Boschat AC, Sanquer S, Guerrera IC, Revy P, Parisot M, Masson C, Boddaert N, Charbit M, Decramer S, Novo R, Macher MA, Ranchin B, Bacchetta J, Laurent A, Collardeau-Frachon S, van Eerde AM, Hildebrandt F, Magen D, Antignac C, van Tilbeurgh H, Mollet G. Nat Commun. 2019 Sep 3;10(1):3967. doi: 10.1038/s41467-019-11951-x. PMID: 31481669 Free PMC article.
Gervason S, Larkem D, Mansour AB, Botzanowski T, Müller CS, Pecqueur L, Le Pavec G, Delaunay-Moisan A, Brun O, Agramunt J, Grandas A, Fontecave M, Schünemann V, Cianférani S, Sizun C, Tolédano MB, D'Autréaux B. Nat Commun. 2019 Aug 8;10(1):3566. doi: 10.1038/s41467-019-11470-9. PMID: 31395877 Free PMC article.
Chevallier ML, Della-Negra O, Chaussonnerie S, Barbance A, Muselet D, Lagarde F, Darii E, Ugarte E, Lescop E, Fonknechten N, Weissenbach J, Woignier T, Gallard JF, Vuilleumier S, Imfeld G, Le Paslier D, Saaidi PL. Environ Sci Technol. 2019 Jun 4;53(11):6133-6143. doi: 10.1021/acs.est.8b06305. Epub 2019 May 22. PMID: 31082212
Kretov DA, Clément MJ, Lambert G, Durand D, Lyabin DN, Bollot G, Bauvais C, Samsonova A, Budkina K, Maroun RC, Hamon L, Bouhss A, Lescop E, Toma F, Curmi PA, Maucuer A, Ovchinnikov LP, Pastré D. Nucleic Acids Res. 2019 Apr 8;47(6):3127-3141. doi: 10.1093/nar/gky1303. PMID: 30605522 Free PMC article.
Evidence of Cis/Trans-Isomerization at Pro7/Pro16 in the Lasso Peptide Microcin J25.
Jeanne Dit Fouque K, Hegemann JD, Zirah S, Rebuffat S, Lescop E, Fernandez-Lima F. J Am Soc Mass Spectrom. 2019 Jun;30(6):1038-1045. doi: 10.1007/s13361-019-02134-5. Epub 2019 Mar 4. PMID: 30834511
Structural Characterization of N-WASP Domain V Using MD Simulations with NMR and SAXS Data.
Chan-Yao-Chong M, Deville C, Pinet L, van Heijenoort C, Durand D, Ha-Duong T. Biophys J. 2019 Apr 2;116(7):1216-1227. doi: 10.1016/j.bpj.2019.02.015. Epub 2019 Feb 26. PMID: 30878202 Free PMC article.
Single-Scan Diffusion-Ordered NMR Spectroscopy of SABRE-Hyperpolarized Mixtures.
Guduff L, Berthault P, van Heijenoort C, Dumez JN, Huber G. Chemphyschem. 2019 Feb 4;20(3):392-398. doi: 10.1002/cphc.201800983. Epub 2019 Jan 4. PMID: 30521115
Abou Samra, A., A. Robert, C. Gov, L. Favre, L. Eloy, E. Jacquet, J. Bignon, J. Wiels, S. Desrat and F. Roussi (2018). "Dual inhibitors of the pro-survival proteins Bcl-2 and Mcl-1 derived from natural compound meiogynin A." Eur J Med Chem 148: 26-38.
Andre, E., V. Derrien, P. Sebban, N. Assrir, E. Lescop and S. Bernad (2018). "Impact of A90P, F106L and H64V mutations on neuroglobin stability and ligand binding kinetics." J Biol Inorg Chem.
Casiraghi, M., M. Damian, E. Lescop, J. L. Baneres and L. J. Catoire (2018). "Illuminating the Energy Landscape of GPCRs: The Key Contribution of Solution-State NMR Associated with Escherichia coli as an Expression Host." Biochemistry 57(16): 2297-2307.
Cavailles, M., A. Bornet, X. Jaurand, B. Vuichoud, D. Baudouin, M. Baudin, L. Veyre, G. Bodenhausen, J. N. Dumez, S. Jannin, C. Coperet and C. Thieuleux (2018). "Tailored Microstructured Hyperpolarizing Matrices for Optimal Magnetic Resonance Imaging." Angew Chem Int Ed Engl 57(25): 7453-7457.
Concilio, M. G., C. Jacquemmoz, D. Boyarskaya, G. Masson and J. N. Dumez (2018). "Ultrafast Maximum-Quantum NMR Spectroscopy for the Analysis of Aromatic Mixtures." Chemphyschem.
Dumez, J. N. (2018). "Spatial encoding and spatial selection methods in high-resolution NMR spectroscopy." Prog Nucl Magn Reson Spectrosc 109: 101-134.
Dumez, J. N. (2018). "Hyperpolarized magnetic resonance in chemistry." Magn Reson Chem 56(7): 565.
Gicquel, M., C. Gomez, M. C. Garcia Alvarez, O. Pamlard, V. Guerineau, E. Jacquet, J. Bignon, A. Voituriez and A. Marinetti (2018). "Inhibition of p53-Murine Double Minute 2 (MDM2) Interactions with 3,3'-Spirocyclopentene Oxindole Derivatives." J Med Chem 61(20): 9386-9392.
Grzela, R., J. Nusbaum, S. Fieulaine, F. Lavecchia, M. Desmadril, N. Nhiri, A. Van Dorsselaer, S. Cianferani, E. Jacquet, T. Meinnel and C. Giglione (2018). "Peptide deformylases from Vibrio parahaemolyticus phage and bacteria display similar deformylase activity and inhibitor binding clefts." Biochim Biophys Acta Proteins Proteom 1866(2): 348-355.
Hamdoun, G., L. Guduff, C. van Heijenoort, C. Bour, V. Gandon and J. N. Dumez (2018). "Spatially encoded diffusion-ordered NMR spectroscopy of reaction mixtures in organic solvents." Analyst 143(14): 3458-3464.
Huber, G., L. Guduff, P. Berthault, C. van Heijenoort and J. N. Dumez (2018). "Single-scan diffusion-ordered NMR spectroscopy of SABRE-hyperpolarized mixtures." Chemphyschem.
Jacquemmoz, C. and J. N. Dumez (2018). "Acceleration of 3D DOSY NMR by Spatial Encoding of the Chemical Shift." Chemphyschem.
Malard, F., N. Assrir, M. Alami, S. Messaoudi, E. Lescop and T. Ha-Duong (2018). "Conformational Ensemble and Biological Role of the TCTP Intrinsically Disordered Region: Influence of Calcium and Phosphorylation." J Mol Biol 430(11): 1621-1639.
Molina, C. E., E. Jacquet, P. Ponien, C. Munoz-Guijosa, I. Baczko, L. S. Maier, P. Donzeau-Gouge, D. Dobrev, R. Fischmeister and A. Garnier (2018). "Identification of optimal reference genes for transcriptomic analyses in normal and diseased human heart." Cardiovasc Res 114(2): 247-258.
Mons, C., T. Botzanowski, A. Nikolaev, P. Hellwig, S. Cianferani, E. Lescop, C. Bouton and M. P. Golinelli-Cohen (2018). "The H2O2-Resistant Fe-S Redox Switch MitoNEET Acts as a pH Sensor To Repair Stress-Damaged Fe-S Protein." Biochemistry 57(38): 5616-5628.
Morellet, N., X. Li, S. A. Wieninger, J. L. Taylor, J. Bischerour, S. Moriau, E. Lescop, B. Bardiaux, N. Mathy, N. Assrir, M. Betermier, M. Nilges, A. B. Hickman, F. Dyda, N. L. Craig and E. Guittet (2018). "Sequence-specific DNA binding activity of the cross-brace zinc finger motif of the piggyBac transposase." Nucleic Acids Res 46(5): 2660-2677.
Mouhand, A., A. Belfetmi, M. Catala, V. Larue, L. Zargarian, F. Brachet, R. J. Gorelick, C. Van Heijenoort, G. Mirambeau, P. Barraud, O. Mauffret and C. Tisne (2018). "Modulation of the HIV nucleocapsid dynamics finely tunes its RNA-binding properties during virion genesis." Nucleic Acids Res 46(18): 9699-9710.
Petitalot, A., E. Dardillac, E. Jacquet, N. Nhiri, J. Guirouilh-Barbat, P. Julien, I. Bouazzaoui, D. Bonte, J. Feunteun, J. A. Schnell, P. Lafitte, J. C. Aude, C. Nogues, E. Rouleau, R. Lidereau, B. S. Lopez, S. Zinn-Justin, S. M. Caputo and U. G. G. B. network (2018). "Combining Homologous Recombination and Phosphopeptide-binding Data to Predict the Impact of BRCA1 BRCT Variants on Cancer Risk." Mol Cancer Res.
Plainchont, B., P. Berruyer, J. N. Dumez, S. Jannin and P. Giraudeau (2018). "Dynamic Nuclear Polarization Opens New Perspectives for NMR Spectroscopy in Analytical Chemistry." Anal Chem 90(6): 3639-3650.
Richard, C. A., V. Rincheval, S. Lassoued, J. Fix, C. Cardone, C. Esneau, S. Nekhai, M. Galloux, M. A. Rameix-Welti, C. Sizun and J. F. Eleouet (2018). "RSV hijacks cellular protein phosphatase 1 to regulate M2-1 phosphorylation and viral transcription." PLoS Pathog 14(3): e1006920.
Rochat, T., C. Bohn, C. Morvan, T. N. Le Lam, F. Razvi, A. Pain, C. Toffano-Nioche, P. Ponien, A. Jacq, E. Jacquet, P. D. Fey, D. Gautheret and P. Bouloc (2018). "The conserved regulatory RNA RsaE down-regulates the arginine degradation pathway in Staphylococcus aureus." Nucleic Acids Res 46(17): 8803-8816.
Romano, M., G. Fusco, H. G. Choudhury, S. Mehmood, C. V. Robinson, S. Zirah, J. D. Hegemann, E. Lescop, M. A. Marahiel, S. Rebuffat, A. De Simone and K. Beis (2018). "Structural Basis for Natural Product Selection and Export by Bacterial ABC Transporters." ACS Chem Biol 13(6): 1598-1609.
Samson, C., A. Petitalot, F. Celli, I. Herrada, V. Ropars, M. H. Le Du, N. Nhiri, E. Jacquet, A. A. Arteni, B. Buendia and S. Zinn-Justin (2018). "Structural analysis of the ternary complex between lamin A/C, BAF and emerin identifies an interface disrupted in autosomal recessive progeroid diseases." Nucleic Acids Res 46(19): 10460-10473.
Tonali, N., J. Kaffy, J. L. Soulier, M. L. Gelmi, E. Erba, M. Taverna, C. van Heijenoort, T. Ha-Duong and S. Ongeri (2018). "Structure-activity relationships of beta-hairpin mimics as modulators of amyloid beta-peptide aggregation." Eur J Med Chem 154: 280-293.
Assrir, N., F. Malard and E. Lescop (2017). "Structural Insights into TCTP and Its Interactions with Ligands and Proteins." Results Probl Cell Differ 64: 9-46.
Dumez, J. N. (2017). "Perspectives on hyperpolarised solution-state magnetic resonance in chemistry." Magn Reson Chem 55(1): 38-46.
Dumez, J. N., B. Vuichoud, D. Mammoli, A. Bornet, A. C. Pinon, G. Stevanato, B. Meier, G. Bodenhausen, S. Jannin and M. H. Levitt (2017). "Dynamic Nuclear Polarization of Long-Lived Nuclear Spin States in Methyl Groups." J Phys Chem Lett 8(15): 3549-3555.
Grzela, R., J. Nusbaum, S. Fieulaine, F. Lavecchia, M. Desmadril, N. Nhiri, A. Van Dorsselaer, S. Cianferani, E. Jacquet, T. Meinnel and C. Giglione (2018). "Peptide deformylases from Vibrio parahaemolyticus phage and bacteria display similar deformylase activity and inhibitor binding clefts." Biochim Biophys Acta Proteins Proteom 1866(2): 348-355.
Guduff, L., A. J. Allami, C. van Heijenoort, J. N. Dumez and I. Kuprov (2017). "Efficient simulation of ultrafast magnetic resonance experiments." Phys Chem Chem Phys 19(27): 17577-17586.
Guduff, L., I. Kuprov, C. van Heijenoort and J. N. Dumez (2017). "Spatially encoded 2D and 3D diffusion-ordered NMR spectroscopy." Chem Commun (Camb) 53(4): 701-704.
Guduff, L., D. Kurzbach, C. van Heijenoort, D. Abergel and J. N. Dumez (2017). "Single-Scan (13) C Diffusion-Ordered NMR Spectroscopy of DNP-Hyperpolarised Substrates." Chemistry 23(66): 16722-16727.
Lopez-Castilla, A., B. Vitorge, L. Khoury, N. Morellet, O. Francetic and N. Izadi-Pruneyre (2017). "(1)H, (15)N and (13)C resonance assignments and secondary structure of PulG, the major pseudopilin from Klebsiella oxytoca type 2 secretion system." Biomol NMR Assign 11(2): 155-158.
Mons, C., I. Ferecatu, S. Riquier, E. Lescop, C. Bouton and M. P. Golinelli-Cohen (2017). "Combined Biochemical, Biophysical, and Cellular Methods to Study Fe-S Cluster Transfer and Cytosolic Aconitase Repair by MitoNEET." Methods Enzymol 595: 83-106.
Munoz-Montesino, C., C. Sizun, M. Moudjou, L. Herzog, F. Reine, A. Igel-Egalon, C. Barbereau, J. Chapuis, D. Ciric, H. Laude, V. Beringue, H. Rezaei and M. Dron (2017). "A stretch of residues within the protease-resistant core is not necessary for prion structure and infectivity." Prion 11(1): 25-30.
Pereira, N., C. Cardone, S. Lassoued, M. Galloux, J. Fix, N. Assrir, E. Lescop, F. Bontems, J. F. Eleouet and C. Sizun (2017). "New Insights into Structural Disorder in Human Respiratory Syncytial Virus Phosphoprotein and Implications for Binding of Protein Partners." J Biol Chem 292(6): 2120-2131.
Rosenberg, J. T., N. Shemesh, J. A. Muniz, J. N. Dumez, L. Frydman and S. C. Grant (2017). "Transverse relaxation of selectively excited metabolites in stroke at 21.1 T." Magn Reson Med 77(2): 520-528.
Rouger, L., M. Yon, V. Sarou-Kanian, F. Fayon, J. N. Dumez and P. Giraudeau (2017). "Ultrafast acquisition of (1)H-(1)H dipolar correlation experiments in spinning elastomers." J Magn Reson 277: 30-35.
Shemesh, N., J. T. Rosenberg, J. N. Dumez, S. C. Grant and L. Frydman (2017). "Distinguishing neuronal from astrocytic subcellular microstructures using in vivo Double Diffusion Encoded 1H MRS at 21.1 T." PLoS One 12(10): e0185232.
Vernhes, E., M. Renouard, B. Gilquin, P. Cuniasse, D. Durand, P. England, S. Hoos, A. Huet, J. F. Conway, A. Glukhov, V. Ksenzenko, E. Jacquet, N. Nhiri, S. Zinn-Justin and P. Boulanger (2017). "Erratum: High affinity anchoring of the decoration protein pb10 onto the bacteriophage T5 capsid." Sci Rep 7: 43977.
Vernhes, E., M. Renouard, B. Gilquin, P. Cuniasse, D. Durand, P. England, S. Hoos, A. Huet, J. F. Conway, A. Glukhov, V. Ksenzenko, E. Jacquet, N. Nhiri, S. Zinn-Justin and P. Boulanger (2017). "High affinity anchoring of the decoration protein pb10 onto the bacteriophage T5 capsid." Sci Rep 7: 41662.
Zargarian, L., A. Ben Imeddourene, K. Gavvala, N. P. F. Barthes, B. Y. Michel, C. A. Kenfack, N. Morellet, B. Rene, P. Fosse, A. Burger, Y. Mely and O. Mauffret (2017). "Structural and Dynamical Impact of a Universal Fluorescent Nucleoside Analogue Inserted Into a DNA Duplex." J Phys Chem B 121(50): 11249-11261.
 La plateforme RMN Très Hauts Champs de Lille comprend les spectromètres RMN 1200 MHz et 900 MHz mixtes liquide-solide, qui sont gérés par l'Unité de Catalyse et de Chimie du Solide (UCCS, UMR 8181 CNRS/Univ. Lille/ENSC-Lille/EC-Lille/Univ. Artois) et l'équipe de recherche mixte Integrative Structural Biology (ISB, EMR 9002 CNRS/INSERM U1167/Univ. Lille). L'UCCS et l'ISB font partie de la fédération FR 2638 (Institut Michel Eugène Chevreul IMEC) qui regroupe les plateformes analytiques en Chimie et Biologie de l'université de Lille.
La plateforme RMN Très Hauts Champs de Lille comprend les spectromètres RMN 1200 MHz et 900 MHz mixtes liquide-solide, qui sont gérés par l'Unité de Catalyse et de Chimie du Solide (UCCS, UMR 8181 CNRS/Univ. Lille/ENSC-Lille/EC-Lille/Univ. Artois) et l'équipe de recherche mixte Integrative Structural Biology (ISB, EMR 9002 CNRS/INSERM U1167/Univ. Lille). L'UCCS et l'ISB font partie de la fédération FR 2638 (Institut Michel Eugène Chevreul IMEC) qui regroupe les plateformes analytiques en Chimie et Biologie de l'université de Lille. RM2I-UCCS :
RM2I-UCCS :
L'équipe de résonance magnétique et matériaux inorganiques (RM2I) de l'UCCS développe et applique des méthodes avancées de RMN, notamment pour les hauts champs magnétiques et les noyaux quadripolaires, afin de caractériser la structure à l'échelle atomique des matériaux inorganiques et hybrides, désordonnés ou amorphes, utilisés dans le domaine de l'énergie (batterie, solaire, hydrogène, nucléaire) et de la catalyse.
ISB : Le groupe de Biologie Intégrative Structurale (ISB) travaille sur la RMN des biomolécules en solution et en solide, en particulier sur les interactions moléculaires (protéine / protéine, protéine / ligand, protéine / lipide) et les modifications post-traductionnelles. Il est reconnu pour le développement de séquences d'impulsions de type Pure-shift appliquées à l'étude de poly peptides.
- Expertises et services
- Solide : haute résolution des noyaux quadripolaires (MQ-MAS, ST-MAS), corrélations hétéro-nucléaires et homo-nucléaires via l'espace ou le couplage J pour des isotopes de spin ½ ou des noyaux quadripolaires (notamment corrélation homonucléaire entre noyaux quadripolaires, corrélation de type HMQC ou INEPT entre isotopes de spin-1/2 et noyaux quadripolaires, y compris entre noyaux de fréquences proches 13C-27Al …), expériences haute-résolution 1H...
- Liquide : RMN des protéines en solutions y compris les protéines non structurée et les protéines de haut poids moléculaire (> 70 kDa par des marquages spécifiques) , corrélations hétéro-nucléaires et homo-nucléaires via l'espace ou le couplage J pour des isotopes de spin ½ 1H-15N-13C études des interactions protéine/protéine ou protéine/ligand (STD, Waterlogsy, CPMG) , étude des mouvements des protéines en solution (relaxation T1/T2, nOe hétéronucléaire 1H-15N, dispersion de relaxation) , spectres 1H homo-découplé (pureshift)
- Autres équipements
Les spectromètres 900 et 1200 MHz s'insèrent dans la plateforme associant les appareils à plus bas champ du pôle RMN de la Faculté des Sciences et Technologies de l'Université de Lille, ceux du Plateau technique de RMN de l'UFR des Sciences de la Santé et du Sport de l'Université de Lille et ceux situés à l'Institut Pasteur. Ces équipements sont mis en commun à travers le Groupement Scientifique RMN de Lille liant les équipes RMN de l'ISB, l'UCCS, le LARMN, le pôle RMN de la Faculté des Sciences et Technologies et le LASIRE.
RMN des solides (Faculté des Sciences et Technologies) :
- 800 MHz SB Avance NEO 3 canaux :
- sondes liquides :
-
BBI 3 mm (1H/BB/2H), z-gradient, Tº : -150° à 180 °C
-
TXI 5 mm (1H/13C/15N/2H), z-gradient, Tº : -50° à 80 °C
-
HRMAS 4 mm (1H/13C/15N/2H), z-gradient, Tº : 2° à 50 °C
-
HRMAS 4 mm (1H/13C/31P/2H), z-gradient, Tº : 2° à 50 °C
-
- sondes solides :
- 1,3 mm DVT HX (1H/14N-31P)
- 3 & 4mm statique PhoenixNMR HX (1H/197Au-71Ga), Q variable, T° : -100° à +150 °C
- 1.6 mm PhoenixNMR HXYD (H=1H-19F, X=31P – 29Si, Y=27Al – 109Ag, lock channel), régulation en T° (-60° à +120 °C), MAS < 40kHz
- 2,5 mm DVT tri-Gamma® HXY (1H / X=31P-13C / Y=15N-29Si avec restrictions de combinaisons)
- 3,2 mm DVT HX (1H/15N–31P)
- 3,2 mm DVT HX (1H/187W–65Cu)
- 3,2 mm VTN PB (31P / 71Ga,11B,115Sn,87Rb), stator VESPEL (sans 11B)
- 3,2 mm PhoenixNMR HXY (H=1H-19F, X=31P – 29Si, Y=27Al – 109Ag), régulation en T° (-30° à +120 °C), MAS < 25kHz
- 3,2 mm DVT tri-Gamma ® HXY (spéciale 1H/13C/14N)
- 3,2 mm DVT tri-Gamma ® HXY (spéciale 1H/11B/29Si)
- 3,2 mm DVT tri-Gamma ® HXY (1H / X=31P-13C / Y=15N-29Si avec restrictions de combinaisons : 29Si seulement avec 31P par exemple)
- 3,2 mm DVT HPX (1H/31P / X=93Nb, 51V-27Al)
- 4 mm HX rotor type Revolution NMR (1H/187W–65Cu ; accord non continu : demander la liste des inserts)
-
Le spectromètre possède une REDORBOX qui, associée à la sonde 3,2 mm ou 1,3 mm HX, permet des corrélations entre noyaux à fréquences proches autour de 200 MHz comme 13C, 27Al.
- sondes liquides :
-
Deux 400 MHz WB (Avance 3 et Avance NEO) 3 canaux :
-
sondes mutualisées sur les deux spectromètres :
-
HXY (1.3mm, 2.5mm, 3.2mm et 4mm) ;
-
HX 4mm (CP standard, bas gamma, WVT et MQMAS) ;
-
HX 7mm bas gamma ;
- HX 5mm statique.
-
-
REDOR Box pour une fréquence nominale de 100 MHz (13C-23Na).
- MAS 2 et 3.
-
RMN des liquides (Faculté des Sciences et Technologies) :
-
400 MHz SB Avance NEO, 3 canaux :
-
sonde BBO 5mm (BB/1H/2H), z-gradient, Tº : -150° à 150 °C ;
-
sonde BBI 5mm (1H/BB/2H), z-gradient, Tº : -150° à 150 °C ;
-
sonde TBI 5mm (1H/13C/BB/2H), z-gradient, Tº : -150° à 150 °C ;
-
sonde BBO 10mm (BB/1H/2H), Tº : -150° à 150 °C.
-
-
300 MHz SB Avance III HD :
-
sonde BBFO 5mm (BB/1H-19F/2H), z-gradient ;
- passeur SampleXpress 60 positions.
-
Institut Pasteur de Lille :
-
600MHz SB Avance III HD :
-
cryosonde QCI 5mm (1H*-19F*/13C*/15N/2H*), z-gradient, Tº : 0° à 80 °C [*:canaux froids] ;
- passeur d'échantillons SampleCase 24 positions.
-
-
300MHz SB Avance III :
-
sonde BBO 5mm (1H-19F/2H/109Ag-31P), z-gradient, Tº : -80° à 120 °C ;
- passeur d'échantillons SampleXpress Lite 16 positions.
-
UFR des Sciences de la Santé et du Sport :
-
500 MHz SB Avance NEO :
-
sonde TXI 5mm (1H/13C/15N/2H), z-gradient ;
-
sonde QNP 5mm (1H-19F/13C/31P/2H), z-gradient ;
-
sonde BBO 5mm (BB/1H/2H), z-gradient ;
-
sonde HRMAS 4mm (1H/13C/2H), z-gradient ;
-
mesure sous éclairage par fibre optique [lampe Hg avec filtres, laser Nd:YAG = 532, 355 et 266 nm].
- passeur d'échantillons 24 positions
-
300 MHz SB Avance NEO :
- sonde BBFO 5mm (BB/1H-19F/2H), z-gradient
-
sonde BBI 5mm (1H/BB/2H), z-gradient ;
-
sonde QNP 5mm (1H-19F/13C/31P), z-gradient ;
- passeur d'échantillons SampleXpress Lite 16 positions.
-
300 MHz SB Avance I :
-
sonde BBFO 5mm (BB/1H-19F/2H), z-gradient ;
-
passeur d'échantillons SampleXpress Lite 16 positions.
-
- 800 MHz SB Avance NEO 3 canaux :
La plateforme RMN du laboratoire CEMHTI (Conditions Extrêmes et Matériaux: Haute Température et Irradiation, CNRS UPR 3079) à Orléans est intégrée à Infranalytics via l'accès à la communauté de son spectromètre RMN 850 MHz (20 T) dédié à la RMN haute résolution solide et à la RMN haute température. Constituée autour d'un parc instrumental de 6 spectromètres RMN (200 → 850MHz), l'équipe MateRMag du CEMHTI possède une expertise internationalement reconnue dans les domaines de :
- La caractérisation et modélisation de la structure des solides organiques, inorganiques ou hybrides et des milieux fondus à l'échelle atomique : matériaux cristallins, amorphes, vitreux de l’industrie verrière ou cimentaire, de la catalyse, dédiés au stockage de l’énergie, biocompatibles, …
- Le suivi des modifications de structure et de mobilité atomique dans les matériaux pour le stockage électrochimique de l'énergie en charge, décharge (ex situ, in situ) : environnement local des atomes, mesure de coefficients de diffusion…
- La quantification de la dynamique des oxydes fondus de très haute température et de son impact sur les propriétés rhéologiques.
- La structure et dynamique de sels fondus à haute température, appliquée aux procédés industriels (électrolyse, nucléaire, métallurgie...).
- Le suivi de procédés (nettoyage, traitement de surface, relâchement par lixiviation), appliqué aux céramiques, verres et vitrocéramiques
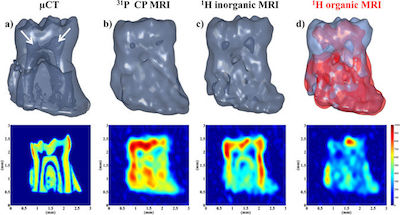
- L'imagerie et spectroscopie localisée dans les matériaux (biomatériaux, milieux poreux pour la catalyse, dispositifs électrochimiques in situ…).
- Expertises et services
- Ordre et désordre structural dans les solides et les milieux fondus, modélisation : matériaux inorganiques, hybrides, cristallins amorphes, vitreux, mésoporeux, biocompatibles...
- Ensemble des techniques standards et avancées de RMN du solide pour la caractérisation des matériaux
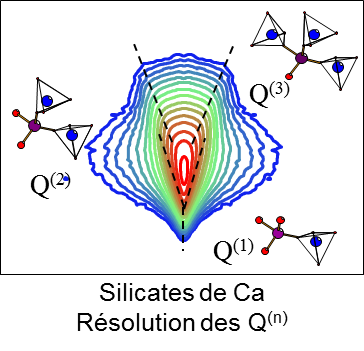
- Noyaux quadripolaires (MQMAS, STMAS)
- Expériences homonucléaires via la liaison chimique ou l'espace (INADEQUATE, BABA, R211, POSTC7...)
- Expériences hétéronucléaires via la liaison chimique ou l'espace (CPMAS, INEPT, HMQC, HSQC, REDOR-type...). Détection indirecte
- Noyaux bas gamma (25Mg, 69Zn, 43Ca, 33S, 73Ge, 14N). Techniques d'amplification du signal (DFS, QCPMG...)
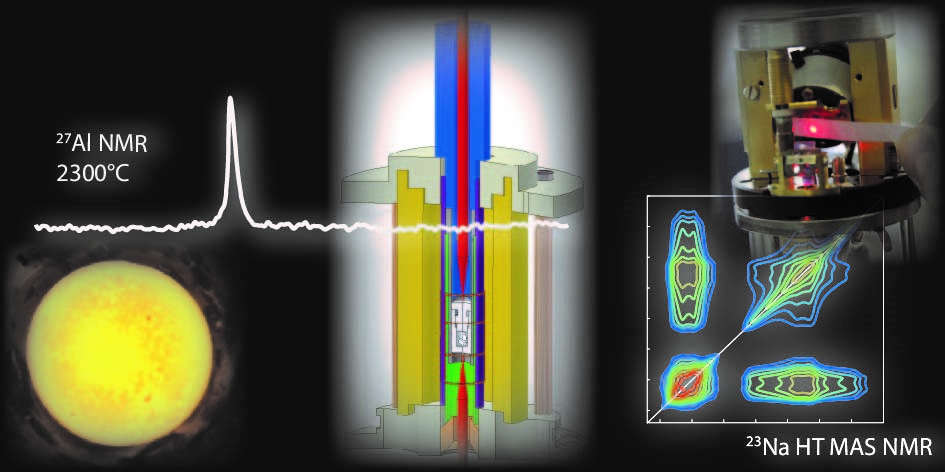
- RMN très hautes températures (conditions statiques et MAS avec chauffage laser)
- Mise à disposition de logiciels : correction de ligne de base "Baseline Corrector" et simulation des spectres "Dmfit" (https://nmr.cemhti.cnrs-orleans.fr/Default.aspx)
- Autres équipements
- 750MHz Bruker Avance III HD solide, haute température, imagerie et diffusion
- 400MHz Bruker Avance III Solide
- 400MHz Bruker Avance I Solide, dédié haute température (chauffage laser)
- 300MHz Bruker Avance III Solide
- 200MHz Bruker Avance III HD Solide, notamment didié à l'étude des matériaux paramagnétiques (dispositifs in situ/operando pour l'electrochimie, sonde MAS 1.3mm 65kHz)
- Autres équipements
600 MHz NB Bruker Avance III Liquide/Solide, « shuttle »
- Sondes liquides :
- TXI (1H, 13C, 15N), 5mm, gradients xyz
- TBI (1H, 13C, X), 5 mm, gradients xyz
400 MHz Bruker Avance III Liquide / Solide
- Sondes liquides:
- TXI (1H, 13C, 15N), 5 mm, gradient-z
- TXI (1H, 13C, 15N), 10 mm
- Sondes solides:
- MAS (1H, X, Y), 1.3 mm
- MAS (1H, X, Y), 2.5 mm
- MAS (1H, X, Y), 4 mm
- MAS (1H, 13C/31P), 4 mm
- MAS (1H, X, Y), 7 mm
A propos de l'IBS
A la fois centre de recherche, plateau technique, site d’accueil et de formation scientifique, l’Institut de Biologie Structurale (IBS) a pour vocation le développement de recherches en biologie structurale, un champ de recherche capital pour la compréhension des mécanismes biologiques fondamentaux. L’IBS s’intègre dans un ensemble plus vaste, le Partenariat pour la Biologie Structurale (PSB ), dont le premier objectif est l’étude des protéines d’intérêt médical.
- Expertises et services
- Fast-NMR (Best, sofast ...)
- RMN en temps réel avec injecteur
- Bio-NMR en phase liquide (simple, double et triple résonances)
- Bio-NMR en phase solide (simple, double et triple résonances)
- Relaxation dipolaire et relaxation dispersion
- RMN des methyls
- Illumination in situ des échantillons par laser
- Autres équipements
850 MHz Bruker Avance III HD Liquide avec passeur d'échantillon
- Sondes liquides
- Cryo TCI (1H, 13C, 15N, 2H), 5 mm, z-gradients
- Cryo TCI (1H, 13C, 15N, 2H), 1,7 mm , z-gradients
700 MHz Bruker Avance III HD Liquide / Solide
- Sonde liquide:
Cryo QCI (1H/31P, 13C, 15N, 2H), 5 mm, z-gradients - Sondes solides:
- MAS (1H, 13C, 15N), 3,2 mm, E-free
- MAS (1H, 13C, 15N), 1,3 mm, +lock
600 MHz (Bruker-1) Bruker Avance III HD LiquideSondes liquides
- Cryo TCI (1H, 13C, 15N, 2H), 5 mm, z-gradients, x,y,z-gradients
600 MHz (Bruker-2) Bruker Avance III HD Liquide / Solide
- Sondes solides:
- MAS (1H, 13C, 15N), 3,2 mm
- MAS (1H, 13C, 15N, 31P), 1,3 mm, +lock
600 MHz (Bruker-3) Bruker Avance III HD Liquide / Solide
- Sonde liquide:
- QCI (1H/19F, 13C, 15N, 2H), 5 mm, z-gradients
- Sondes liquides



